„Wie oft sich Proteine begegnen, ist sehr relevant“
Denise Müller-Dum
Marie Abhag Louise/Universität Siegen
Proteine spielen eine wichtige Rolle für unser Leben: Sie erfüllen eine Vielzahl von Funktionen, darunter die Katalyse von Stoffwechselreaktionen, die Strukturierung von Zellen und Organismen und den Transport von Molekülen. Doch auch einige Krankheiten – wie etwa Alzheimer oder Parkinson – werden durch Proteine verursacht. Um das Verhalten von Proteinen in biologischen Umgebungen besser zu verstehen, haben Forschende nun mithilfe von Röntgenstrahlung untersucht, was beim Kochen von Eigelb passiert. Im Interview erklärt Christian Gutt von der Universität Siegen, welche weiteren Fragen sie in Zukunft mit dieser Methode klären möchten.
Welt der Physik: Wie sind Sie dazu gekommen, Eigelb zu untersuchen?
Christian Gutt: Wir wollen verstehen, wie sich Proteine in hochdichten Lösungen bewegen – also in Umgebungen, die sehr vollgepackt sind mit Proteinen. Solche hochdichten Lösungen findet man in der Biologie häufig, etwa in unseren Körperzellen oder auch in Lebensmitteln. In unserem Projekt untersuchen wir, wie sich die Proteine in solchen Lösungen bewegen. Hühnereier sind dafür ein ideales Modellsystem: Sie enthalten viele Proteine, sind leicht zu besorgen, günstig und einfach zu handhaben. Wir haben vor ein paar Jahren schon Hühnereiweiß untersucht und jetzt eben Eigelb – was sich nochmal als spannender herausgestellt hat, weil es eine besondere innere Struktur hat.
Woraus besteht denn das Eigelb von Hühnereiern?
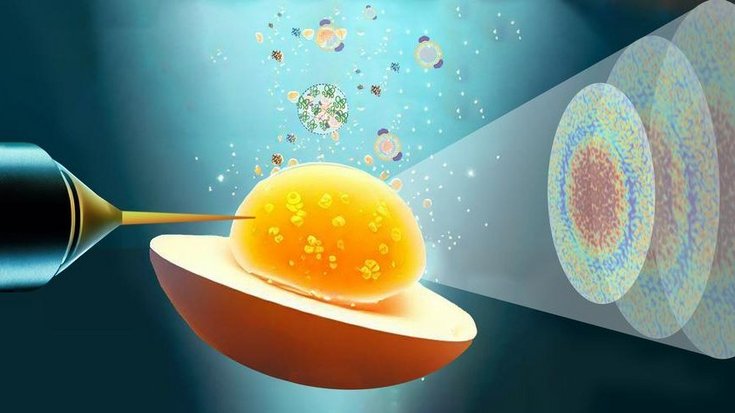
Zum einen besteht es aus vielen verschiedenen Proteinen und zum anderen aus kleinen kugelförmigen Fett-Eiweiß-Teilchen, die wir LDL nennen und die unter anderem das Cholesterin transportieren. Diese LDL haben einen Durchmesser von 20 bis 30 Nanometern. Außerdem liegen 800 Nanometer bis ein Mikrometer große Partikel im Eigelb vor, die sogenannten Granula. Wenn man das Ei kocht, verändert sich die gesamte Struktur: Die Proteine, die normalerweise gefaltet sind, öffnen sich – sie denaturieren. Sie verhalten sich dann wie doppelseitiges Klebeband und haften mit allem, was sie umgibt, zusammen und bilden Aggregate. Dann verschmelzen die Fettkügelchen miteinander, und am Schluss auch die großen Granula. Deswegen wird das Eigelb so körnig, wenn man es sehr lange kocht.
Was interessiert Sie an dieser Strukturveränderung?
Es ist natürlich für sich genommen schon spannend, wie Proteine sich bewegen und Mikrostrukturen formen. Es gibt beim Menschen aber zudem einige Krankheiten, die mit der Denaturierung oder Aggregation von Proteinen im Zusammenhang stehen. Einer der diskutierten Mechanismen bei Alzheimer ist zum Beispiel, dass Proteine sich zuerst zu Ketten und schließlich zu größeren Bündeln zusammenschließen, die dann die Nervenzellen absterben lassen. Wie oft sich Proteine begegnen, um überhaupt solche Aggregate bilden zu können, ist also eine wichtige Frage – deshalb interessieren wir uns so sehr für die Dynamik von Proteinen.
Wie haben Sie die Dynamik der Proteine im Eigelb untersucht?
Wir haben das Eigelb mit einer Spritze aufgezogen und in dünne Röhrchen gefüllt. Diese haben wir dann in ein Wasserbad gestellt und erhitzt und den Prozess zeitaufgelöst mit Röntgenlicht beobachtet. Für diese Experimente hatten wir mehrere Messzeiten an der Synchrotronquelle PETRA III am Forschungszentrum DESY in Hamburg.
Und wie genau haben Sie mit dem Röntgenlicht das Eigelb untersucht?
Wenn man kohärentes, also laserartiges, Röntgenlicht auf eine Probe schickt, wird das Röntgenlicht an den Bestandteilen der Probe gebeugt. Und dieses gebeugte Röntgenlicht können wir beobachten. Ist die Probe ungeordnet, dann ergibt sich zum Beispiel ein diffuses Bild mit vielen Tupfen. In unserem Fall lenken die großen Aggregate das Licht eher unter einem kleinen Winkel ab. Und an den kleineren Fettmolekülen und Proteinen wird es mit größeren Winkeln gestreut, wodurch weiter außen auf dem Kamerabild Tupfen entstehen. Wenn sich die Probe verändert, dann fangen auch diese Tupfen an sich zu bewegen. Diese Bewegung sagt also etwas über die Dynamik der Bestandteile der Probe aus. Indem wir alle 100 Mikrosekunden ein Bild aufnehmen, erhalten wir einen komplizierten Film, den wir aufwendig analysieren und so etwas über die Prozesse im Eigelb erfahren.
Was haben Sie über diese Prozesse herausgefunden?
Was wir gesehen haben, ist: Ganz gleich, bei welcher Temperatur man das Eigelb kocht – die Prozesse, die stattfinden, sind im Prinzip immer die gleichen. Sie laufen nur unterschiedlich schnell ab, je nachdem, ob ich eine niedrige Temperatur habe oder eine hohe Temperatur. In der Sprache der Physik sagt man: Das Verhalten der sehr unterschiedlichen und komplexen Bestandteile des Eigelbs gehorcht einfachen Skalengesetzen. Das ist eine tolle Sache: Denn wenn sich selbst komplexe Systeme in bestimmter Hinsicht durch einfache Modelle beschreiben lassen, dann gibt uns das ein mächtiges Werkzeug an die Hand, um tiefere Einsichten in die Natur zu gewinnen. Es erlaubt uns sogar, unsere Ergebnisse auf Temperaturbereiche zu übertragen, die wir gar nicht angeschaut haben, wie etwa Körpertemperatur. Das war eine überraschende und erfreuliche Erkenntnis. Außerdem haben wir ein sogenanntes Phasendiagramm aufgestellt. Damit können wir uns genau anschauen, welche Prozesse im Eigelb – abhängig von der Zeit und der Temperatur – beim Kochen ablaufen. Das gab es bislang noch nicht. Mit der Übertragung in die Küche muss man allerdings vorsichtig sein: Wir haben uns ja nur Eigelb in einem dünnen Röhrchen angeschaut. Das Verhalten eines richtigen Eis ist noch etwas komplizierter.
Und was wollen Sie als Nächstes erforschen?
Nach der Veröffentlichung unserer Ergebnisse haben Forschende angefragt, ob wir auch kompliziertere biologische Proben mit unserer Methode untersuchen können – unter anderem die Proteine, die beim Coronavirus eine Rolle spielen, oder eben bei Alzheimer. Das hat mich ziemlich gefreut und ist auf jeden Fall etwas, das wir uns in Zukunft anschauen wollen. Eine weitere Sache, die ich spannend finde, sind monoklonale Antikörper – also moderne Medikamente, die nur aus Proteinen bestehen. Die müssen Sie sich als hochdichte Lösung, sozusagen wie Honig, vorstellen. Da solche Medikamente noch sehr schwierig zu injizieren sind, stellt sich die Frage, warum diese hochdichten Proteinlösungen überhaupt so zähflüssig sind? Und zum anderen: Lassen sich Methoden finden, um die Medikamente weniger viskos zu machen? Daran hat die Pharmaindustrie großes Interesse.
Gibt es etwas an Ihrer Methode, das Sie in Zukunft weiterentwickeln möchten?
Ja, wir wollen zum Beispiel die Wechselwirkung zwischen Röntgenstrahlen und Proteinen genauer untersuchen. Röntgenlicht schädigt die Proteine, und das müssen wir in den Griff bekommen, weil es den beobachteten Vorgang selbst beeinflusst. Dazu müssen wir verstehen, wann eine Dynamik sozusagen künstlich mit dem Röntgenstrahl ausgelöst wird und wann man wirklich die Dynamik in der Probe beobachtet. Da brauchen wir noch mehr Experimente. Die zweite Herausforderung sind die Datenmengen. Denn wenn wir viele von diesen Filmen aufnehmen, haben wir sehr, sehr viele Daten. Hier wollen wir in Zukunft maschinelles Lernen einsetzen, um schon während der Messung eine Rückmeldung zum Erfolg des Experiments zu bekommen.
Das Bundesministerium für Bildung und Forschung fördert den Verbund „Protein-Dyn: Dynamik von Proteinen in Lösungen auf multiplen Längen und Zeitskalen“ im Zeitraum von Juli 2020 bis Juni 2024 mit rund 900 000 Euro.
Fördersumme: 903 132 Euro
Förderzeitraum: 01.07.2020 bis 30.06.2024
Förderkennzeichen: 05K20PSA, 05K20VTA
Beteiligte Institutionen: Universität Siegen, Universität Tübingen
Quelle: https://www.weltderphysik.de/thema/bmbf/erforschung-kondensierter-materie/petra-iii-wie-oft-sich-proteine-begegnen-ist-sehr-relevant/